Vacunas contra Covid 19
Diciembre - 2020
Targets virales para las vacunas contra COVID-19
Lianpan Dai and George F. Gao
CAS
Key Laboratory of Pathogenic Microbiology and Immunology, Institute of
Microbiology, Beijing, China.
Nature Reviews | Immunology 2020 Published online 18 - 12 -2020
Traducción libre Dr Gerardo Flores H Pediatra Neonatólogo Hospital Puerto Montt
Al 3 de diciembre de 2020, la
pandemia de la enfermedad por coronavirus 2019 (COVID-19), causada por
la infección por Sindrome respiratorio agudo severo - coronavirus 2 (SARS-CoV-2)
, se ha extendido a 220 países, áreas o territorios con más de 63
millones de casos confirmados por laboratorio y más de 1.4 millones de
muertes (Organización Mundial de la Salud (OMS) ) , lo que ha provocado
trastornos sociales y económicos generalizados. Se necesita con urgencia
el desarrollo de una vacuna segura y eficaz para ayudar a poner fin a
esta pandemia.
El SARS-CoV-2 es un miembro de la familia Coronaviridae, que comprende
muchas cepas virulentas que infectan a seres humanos y animales,
incluyendo el SARS-CoV y el síndrome respiratorio CoV de Oriente Medio (MERS-CoV)
1. Hasta la fecha, se han autorizado varias vacunas CoV para
su uso en animales domésticos contra CoV canino, CoV felino, CoV bovino
(BCoV), virus de la diarrea epidémica porcina, virus de la
gastroenteritis transmisible (TGEV) y virus de la bronquitis infecciosa
(IBV) 2; sin embargo, hasta ahora, ninguna ha sido autorizada
para su uso en humanos. Dos candidatas a vacunas para SARS-CoV y tres
para el MERS-CoV se encuentran en ensayos clínicos de fase I
(OMS). Estas experiencias previas de desarrollo de vacunas para CoV
animales y humanos han proporcionado información importante sobre el
desarrollo de vacunas para la infección por SARS-CoV-2. Para desarrollar
una vacuna contra un virus de reciente aparición, es importante
comprender los correlatos inmunitarios de la protección.
Aunque queda mucho por determinar con respecto a los correlatos inmunitarios de protección para la infección por SARS-CoV-2, datos emergentes han demostrado la importancia de la inmunidad tanto humoral como celular en la protección. Se ha encontrado una fuerte correlación entre los anticuerpos neutralizantes inducidos por la vacuna (nAbs) y una reducción de las cargas virales en primates no humanos (NHPs) después de la infección por SARS-CoV-23–6.
En humanos, se ha reportado que la administración pasiva de plasma de convalescientes 7-10, IgG purificada11,12 o anticuerpos monoclonales13 muestra beneficios para el tratamiento y la prevención de la infección por SARS-CoV-2. En particular, un nAb recibió recientemente la autorización de la Administración de Alimentos y Medicamentos de EE. UU. (FDA) para uso de emergencia como tratamiento para COVID-19 (referencia 14). Además, el análisis de un brote de COVID-19 a bordo de un barco pesquero con altas tasas de infección apoyó la correlación de nAbs con protección15. Además de los nAbs , las respuestas de las células T también desempeñan roles protectores críticas en las infecciones por CoV. Se ha demostrado que la depleción de células T en ratones altera el clearance o eliminación del virus en las infecciones por SARS-CoV, MERS-CoV y SARS-CoV-216-19. En los pacientes, las respuestas de las células T CD4 + y CD8 + virus específicas se asocian con una enfermedad más leve, lo que sugiere una participación en la inmunidad protectora contra COVID-19. 20-22 Por lo tanto, se espera que una vacuna ideal evoque las ramas humoral y celular del sistema inmune.
Sin embargo, una preocupación de seguridad importante para el desarrollo de una vacuna contra el SARS-CoV-2 o de terapias basadas en anticuerpos es el riesgo potencial de que :
-
La vacuna facilite la enfermedad , lo que también se conoce como facilitamiento dependiente de anticuerpos (ADE) un proceso en el que un virus aprovecha los anticuerpos para ayudar a la infección los anticuerpos se fijan al virus, hacen complejos inmunes son fagocitados y dentro de los macrófagos generan un buen sitio para reproducirse eludiendo la inmunidad innata e inespecífica y
-
La enfermedad respiratoria facilitada (enhancement) (ERD). ) 23 mejora basada en células, una categoría que incluye inflamación alérgica causada por inmunopatología Th2, que genera una potenciación de una enfermedad respiratoria asociada a la vacuna "associated enhanced respiratory disease" o respuesta inflamatoria alérgica con respuestas predominantemente celulares que resultan en una disfunción aérea y un retraso en el aclaramiento del virus.
Los anticuerpos que se pueden unir a un virus sin actividades neutralizantes pueden causar ADE a través de la captación del virus mediada por el receptor Fcγ, lo que permite la replicación posterior del virus o funciones efectoras mediadas por Fc del complejo inmune anticuerpo-virus, permitiendo la inmunopatología23,24. Este efecto se asocia típicamente con flavivirus, tales como el virus del dengue25,26 y el virus Zika27, pero también se ha descrito en la infección por CoV. Los gatos inmunizados con el virus vaccinia que expresaba una proteína viral del virus de la peritonitis infecciosa felina (FIPV; un CoV felino) o anticuerpos administrados pasivamente con anticuerpos anti-FIPV mostraron una mortalidad temprana cuando fueron expuestos al virus vivo28-31. También se observó ADE para SARS-CoV y MERS-CoV en modelos animales32-37. Además del ADE, la intensificación de la enfermedad inducida por la vacuna también puede ser causada por inmunopatología sesgada por células T helper 2 (TH2), lo que causa ERD38 - 41.
Aunque algunos estudios del SARS-CoV en modelos animales no muestran evidencia de ADE o ERD33,42,43, se debe considerar la seguridad al diseñar vacunas para el SARS-CoV-2. Con los casos continuos y las muertes por la pandemia de COVID-19, los investigadores de todo el mundo están compitiendo para desarrollar vacunas contra COVID-19.
Según el documento de panorama de la OMS, las candidatas a vacunas COVID-19 generalmente se dividen en siete estrategias (Cuadro 1), que se pueden dividir en tres categorías amplias44 :
-
Primero, vacunas basadas en proteínas que generan antígenos target o diana in vitro, tales como las vacunas de virus inactivados , vacunas de subunidades de proteínas y partículas similares a virus
-
En segundo lugar, vacunas basadas en genes que suministran genes que codifican antígenos virales a las células hospedadoras para la producción in vivo, tales como vacunas vectorizadas por virus, vacunas de ADN y vacunas de ARNm; y
-
Tercero, una combinación de ambos enfoques tanto basados en proteínas como basados en genes para producir antígenos o antígenos proteicos tanto in vitro como in vivo, representadas típicamente por vacunas de virus vivos atenuados.
Hasta diciembre de 2020, la OMS ha
documentado más de 214 candidatos a vacunas COVID-19, 51 de
ellos en evaluación clínica, 13 en ensayos de fase III y varias vacunas
están siendo autorizadas para su uso en algunas regiones (OMS; Rastreador de
vacunas COVID-19 ).
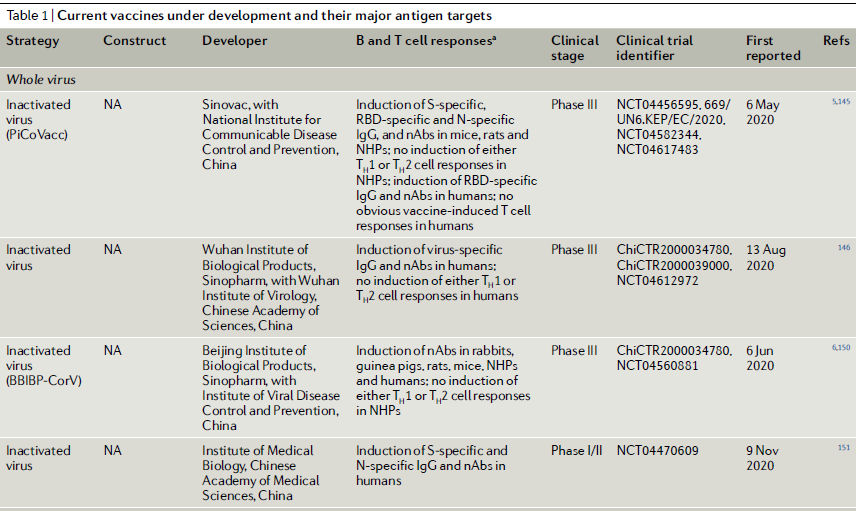
Tabla 1 | Estrategias de Vacunas candidatas para SARS- CoV-2
Vacunas de virus inactivados
Los virus so físicamente ó químicamente inactivados pero preservan la integridad de la partícula viral, la cual sirve como el inmunógeno.
Vacunas de partículas o nanopartículas similares a virus
En esta estrategia, proteínas virales estructurales se coexpresan para formar partículas no infecciosas como inmunógeno de la vacuna.
Se asemejan a viriones reales pero carecen del genoma del virus.
Vacunas de subunidades proteicas
Esta estrategia comprende solo péptidos o proteínas virales clave que pueden fabricarse in vitro en células de bacterias, levaduras, insectos o mamíferos.
El mayor número de candidatas a vacuna contra Sars-Cov-2 tanto en la etapa clínica como preclínica, se basa en esta estrategia.
Vacunas vectorizadas por virus
El gen o genes que codifican los antígenos patógenos se clonan en vectores de virus que no se replican o que se replican (como por ejemplo el adenovirus).
El o los antígenos son producidos por transducción por célula huésped después de inmunización.
Vacunas DnA y mRnA
Las vacunas DNa y mrNa tienen la ventaja de una fabricación rápida contra patógenos emergentes.
Para las vacunas de DNa, los antígenos virales codificados por un plásmido de DNa recombinante se producen en las células huésped mediante un proceso secuencial de transcripción a transducción.
Por el contrario, las vacunas de mrNa se sintetizan mediante transcripción in vitro y producen antígenos virales en el citoplasma mediante la transducción directa de proteínas in vivo.
Vacunas de virus vivos atenuados
En esta estrategia, el virus se atenúa mediante pases in vitro o in vivo o mutagénesis genética inversa.
El virus resultante se vuelve no patógeno o débilmente patógeno pero conserva la inmunogenicidad imitando la infección por virus vivos.
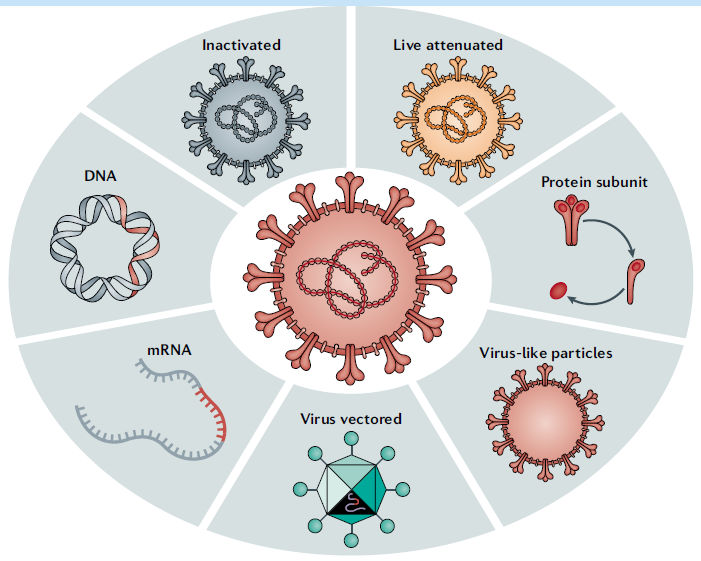
En este artículo de Progress, los autores resumen y discuten los targets u objetivos usados en las candidatas a vacunas, centrándose en aquellas candidatas que ya han avanzado en ensayos clínicos y con datos publicados.
Proteínas SARS-CoV-2 como Targets
El SARS-CoV-2 contiene cuatro proteínas
estructurales principales, llamadas :
-
Proteínas spike o espiga (S)
-
Membrana (M)
-
Envoltura (E),
-
Nucleocápside (N)
todas las cuales están incrustadas en la envoltura de la superficie viral, a lo largo del genoma del ARN o RNA viral. Además de estas proteínas estructurales, el SARS-CoV-2 codifica 16 proteínas no estructurales (nsp 1-16) y 9 proteínas accesorias. Varias de estas proteínas virales podrían potencialmente servir como objetivos o targets de las respuestas inmunes inducidas por vacunas.
Proteína S.
La proteína S es la principal proteína utilizada como target en las vacunas COVID-19. La proteína S consta de una subunidad S1 distal a la membrana y una subunidad S2 proximal a la membrana y existe en la envoltura del virus como un homotrímero. La subunidad S1 determina el reconocimiento del receptor a través de su dominio de unión al receptor (RBD), mientras que la subunidad S2 es responsable de la fusión de membranas, la cual es necesaria para la entrada del virus (Figura 1).
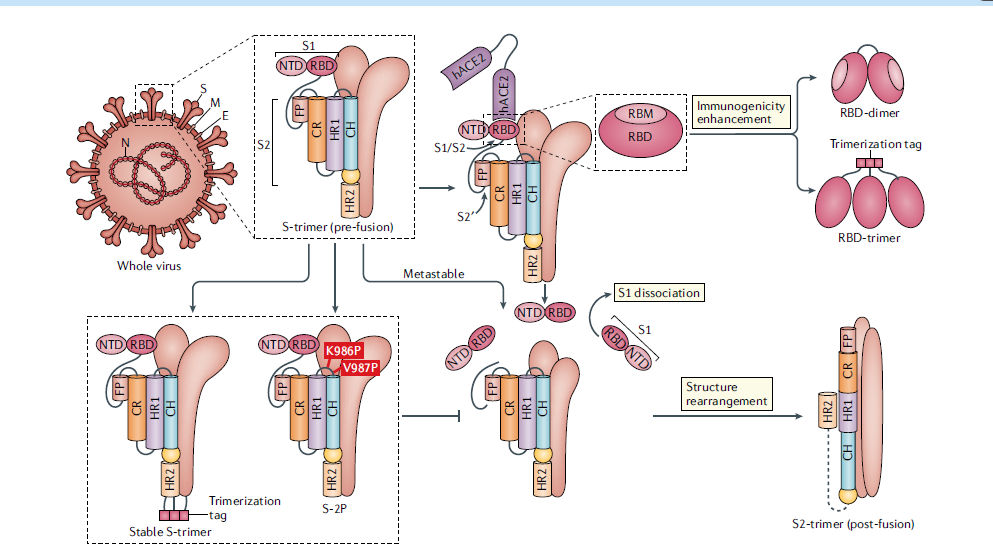
Figura 1.- Principales targets utilizados en candidatas a vacuna CoVID-19. El coronavirus 2 - Síndrome respiratorio agudo severo (SARS-CoV-2) contiene cuatro proteínas de estructura : proteína spike o espiga (S), membrana (M) y envoltura (E), que están incrustadas en la superficie del virión y proteína de la nucleocápside (N). que se une al ARN viral dentro del virión. Se muestra el trímero de la proteína S en su conformación previa a la fusión. La proteína S comprende la subunidad S1 (que incluye el dominio N-terminal (NTD) y el dominio de unión al receptor (RBD)). (El motivo de unión al receptor (RBM) dentro del RBD también está marcado) y la subunidad S2 (que incluye péptido de fusión (FP), región de conexión (CR), repetición de heptada 1 (HR1), repetición de heptada (HR2) y hélix central (CH)). La proteína SARS-CoV-2 S se une al receptor del huésped, la enzima convertidora de angiotensina humana dimérica 2 (hACE2), a través de la RBD y disocia las subunidades S1. La escisión en los sitios S1 – S2 y S2 ′ permite el reordenamiento estructural de la subunidad S2 necesario para la fusión de la membrana del huésped con el virus. Se muestra el trímero S2 en su disposición posterior a la fusión. El RBD es un target de vacuna atractivo. Se ha demostrado que la generación de un dímero RBD o un trímero RBD mejora la inmunogenicidad de las vacunas basadas en RBD. Un trímero S estabilizado que se muestra con una etiqueta de trímero C-terminal es un target de la vacuna. La proteína S previa a la fusión generalmente es metaestable durante las preparaciones in vitro y es propensa a transformarse en su conformación posterior a la fusión. La mutación de dos residuos (K986 y V987) en prolina estabiliza la proteína S (S-2P) y previene el cambio estructural de pre-fusión a post-fusión.
En MERS-CoV, SARS-CoV o SARS-CoV-2,
el RBD se encuentra en el dominio C-terminal de la subunidad S1
46–48. En algunos CoV, el dominio N-terminal (NTD) de la subunidad
S1 puede usarse para la unión al receptor (tal como en el virus de la
hepatitis de ratón) o también podría estar involucrado en la unión del
virus a las células huésped mediante el reconocimiento de moléculas de
azúcar específicas (tal como en TGEV, BCoV e IBV) o tiene un papel
importante en la transición de pre-fusión a post-fusión de la proteína S49-51.
La subunidad S2 contiene el péptido de fusión (FP), la región de
conexión (CR), la repetición 1 de la heptada (HR1) y la HR2 alrededor de
una hélix central como una estructura de hélice-vuelta-hélice (Figura
1). La evidencia estructural
ha propuesto un modelo para el reordenamiento de la proteína S del SARS-CoV-2
después del reconocimiento del receptor de la célula huésped52,53.
La proteína S se puede escindir proteolíticamente en los sitios de
escisión S1-S2 y S2 ′, liberando las limitaciones estructurales en el FP
(Figura 1).
La unión del RBD con su receptor celular, la enzima convertidora de
angiotensina humana 2 (hACE2), causa disociación de la subunidad S1 y
concomitantemente inicia el proceso de replegamiento de la subunidad S2
cargada por resorte, que sobresale el FP en su extremo para fusión de
membranas.
La subunidad S2 en su conformación
post-fusión se pliega como un paquete helicoidal largo con el FP
insertado en la membrana de la célula huésped (Figura
1). En teoría, los nAbs pueden
dirigirse a la proteína S para inhibir la infección por virus en
múltiples etapas durante el proceso de entrada del virus. El RBD es el
principal objetivo de los nAb que interfieren con la unión del receptor
viral54–56. Hasta la fecha, la mayoría de los nAb potentes
contra el SARS-CoV-2 se dirigen al RBD57–69. Además, se han
reportado nAbs dirigidos a las ETD en la infección por SARS-CoV-2 y MERS-CoV49,63,70,71,
lo que la convierte en otro target u objetivo potencial para su
inclusión en una vacuna.
La subunidad S2 también es un target potencial para nAbs que interfiere
con el reordenamiento estructural de la proteína S y la inserción de FP
necesarios para la fusión de la membrana del huésped con el virus49,71-73.
La proteína S también es un target para las respuestas de las células T;
los estudios de SARS-CoV, MERS-CoV y SARS-CoV-2 han descrito epítopes de
células T CD4 + y CD8 + en la proteína S74,75.
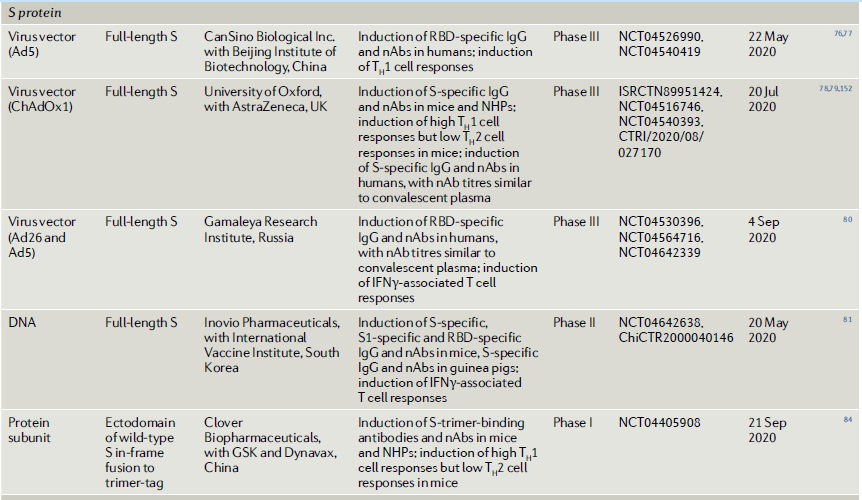
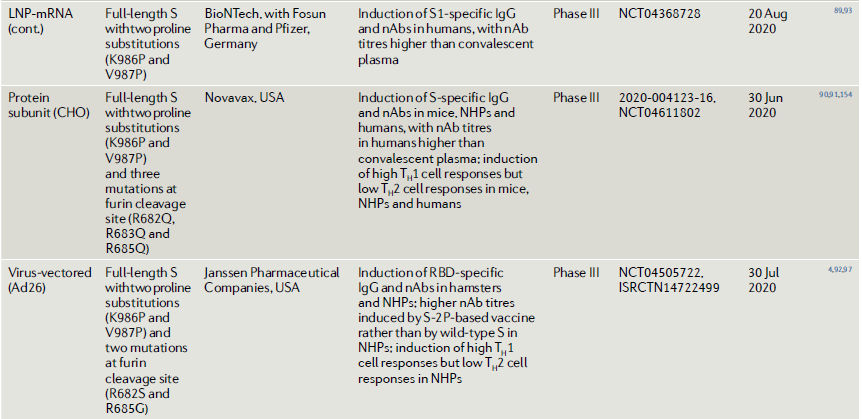
Hasta la fecha, tres candidatas a vacuna COVID-19 basadas en adenovirus que expresan la proteína S de longitud completa han entrado en ensayos clínicos de fase III (Tabla 1).
-
Uno, que se está desarrollando en China, se basa en el adenovirus humano tipo 5 (Ad5) 76,77.
-
El segundo, que se está desarrollando en el Reino Unido, usa adenovirus de chimpancé recombinante, ChAdOx1 78,79.
-
La tercera vacuna candidata, de Rusia, combina Ad26 y Ad5 humanos recombinantes en un régimen de vacunación de refuerzo80.
Los datos preclínicos mostraron que la vacuna ChAdOx1 recombinante inducía la producción de nAb y equilibraba el TH1 TH2 en los NHP78 (Tabla 1). La vacuna previno la neumonía asociada al COVID-19 en los NHP y redujo la carga viral tanto en el líquido de lavado broncoalveolar como en el tracto respiratorio78. Los estudios de ensayos de fase II mostraron que todos estos candidatos a vacunas indujeron la producción de nAb y respuestas sesgadas por las células TH1 en humanos76,79,80 (Tabla 1)
Además de la estrategia de vector de adenovirus, un candidato a vacuna de ADN que expresa la proteína S también ha completado los ensayos clínicos de fase II. Esta vacuna provocó respuestas de células T específicas de la proteína nAbs y S tanto en ratones como en cobayas81 (Tabla 1).
Recientemente, se reportó una proteína S trimérica similar a la nativa para una candidata a vacuna de la subunidad COVID-19 que se está desarrollando en China, en la que la proteína S se fusiona con una etiqueta trímera, la región C-terminal del colágeno humano de tipo Iα, para formar un homotrímero con enlaces disulfuro82. Esta estrategia estabiliza el antígeno de la proteína S en su forma trimérica y aumenta el rendimiento de antígeno82.
La proteína trimérica recombinante imita estructuralmente la proteína S nativa presentada en las partículas del virus y podría servir como target para nAbs, una estrategia que también se está siguiendo en el desarrollo de una vacuna contra el VIH83.
La vacunación del trímero S junto
con AS03 (un adyuvante a base de escualeno desarrollado por
GlaxoSmithKline) o CpG 1018 (un adyuvante agonista del receptor 9 tipo
Toll desarrollado por Dynavax) provocó altos niveles de nAbs y
respuestas sesgadas por células TH1 en ratones y NHP . Podría proteger a
los NHP del desafío con el SARS-CoV-2, con cargas virales y patología
pulmonar disminuídas84. Esta vacuna candidata se encuentra
actualmente en evaluación en ensayos clínicos de fase I (Tabla
1). La proteína S es
metaestable cuando se produce como una proteína recombinante y es
propensa a transformarse de su conformación pre-fusión a una
conformación post-fusión, desprendiendo la subunidad S1 (Figura
1).
Tabla 1.- Vacunas en desarrollo y sus principales antígenos targets (Ver Tabla colocada más abajo antes de Referencias)
Sin embargo, la subunidad S1
es el antígeno inmunodominante durante las infecciones por CoV debido a
su accesibilidad para el reconocimiento inmunológico y contiene epítopes
neutralizantes principalmente en su RBD. Se cree que las estrategias
para estabilizar la proteína S en su conformación previa a la fusión y
mejorar la expresión de la proteína S previa a la fusión aumentan la
calidad y cantidad de anticuerpos inducidos por la vacuna que se dirigen
a los epítopes funcionalmente relevantes en la subunidad S1. Pallesen y
col. reportaron dos sustituciones de prolina (2P) en el vértice de la
hélice central y HR1 que pueden retener las proteínas S de MERS-CoV,
SARS-CoV y HKU1 en la conformación de pre-fusión óptima antigénicamente85
(Figura 1).
El antígeno resultante, S-2P,
indujo títulos de nAb mucho mayores que la proteína S de tipo salvaje en
ratones. Aprendiendo de la experiencia anterior con estos CoV, el diseño
S-2P se está utilizando ahora en varias estrategias de vacuna contra
COVID-19. La SRAS-CoV-2 S-2P (que comprende sustituciones de
prolina en los residuos K986 y V987) se utiliza como antígeno diana en
tres candidatas a vacunas basadas en genes (vacunas de ARNm de Moderna /
Instituto Nacional de Alergias y Enfermedades Infecciosas (NIAID) y
BioNTech / Pfizer y una vacuna Ad26 recombinante de Janssen
Pharmaceutical Companies) y una candidata a base de proteínas (de
Novavax) 4,86–96 (Tabla 1).
Además, también se cree que la mutación en los sitios de escisión en la
proteína S estabiliza la conformación previa a la fusión de la proteína
S. S-2P en la vacuna con vector de Janssen Ad26 (Ad26.COV2.S) y en
la vacuna basada en proteínas Novavax (NVX-CoV2373) contiene mutaciones
adicionales en el sitio de escisión polibásico S1-S2 de RRAR a SRAG o
QQAQ para convertirlo resistente a proteasa, lo que ayuda a estabilizar
aún más la proteína S en su conformación previa a la fusión96,97.
Todos estas candidatas obtuvieron
inmunidad protectora en modelos animales86,87,90 y han
avanzado a ensayos clínicos de fase III (Tabla 1). En particular, un
estudio preclínico mostró que el Ad26 que expresa S-2P provocó títulos
de nAb más altos y una mejor protección en los NHP que otros candidatos
basados en la proteína S de tipo salvaje4. Los estudios
clínicos para las dos vacunas de ARNm (BNT162b2 de BioNTech / Pfizer y
ARNm1273 de Moderna / NIAID) y la vacuna de subunidad de proteínas, NVX-CoV2373,
mostraron de manera alentadora que todas estas vacunas produjeron
títulos altos de nAb más allá de los niveles observados en pacientes
convalecientes y respuestas sustanciales de células T 88,89,91,93.
Por tanto, la estabilización de la proteína S en su conformación previa
a la fusión representa una vía eficaz para mejorar la eficacia de la
vacuna. En particular, aunque los datos aún no se han publicado, los
comunicados de prensa recientes de BioNTech / Pfizer y Moderna / NIAID
han anunciado que sus vacunas de ARNm tienen una eficacia superior al
90% en la prevención de COVID-19 en ensayos clínicos de fase III
98,99. El Reino Unido acaba de aprobar la vacuna BioNTech / Pfizer100.
Trás sus observaciones iniciales en MERS-CoV, SARS-CoV y SARS-CoV-2,
Hsieh et al. generaron una nueva forma variante del ectodominio de la
proteína S, conocida como HexaPro, que comprende seis sustituciones de
prolina beneficiosas, incluidas las dos de S-2P 101. Estas
sustituciones de prolina están ubicadas en bucles flexibles o extremos N
de hélices en FP, HR1 y CR, lo que limita aún más el reordenamiento
estructural de la subunidad S2 y estabiliza la proteína S de prefusión.
HexaPro exhibe una expresión aproximadamente 10 veces mayor que S-2P, lo
que representa un diseño de antígeno prometedor101.
El RBD.
El RBD se une al receptor del
huésped a través de unión al receptor (RBM) en su subdominio externo en
SARS-CoV, MERS-CoV o SARS-CoV-2 47,50,102. La superficie de
la proteína S está ampliamente protegida del reconocimiento de
anticuerpos por los glucanos, con la notable excepción del RBD, que
explica la inmunodominancia de los epítopos del RBD103. La
mayoría de los nAb de SARS-CoV-2 se unen a RBD y bloquean la interacción
RBD-hACE2, inhibiendo así la unión del virus57–69.
La RBD es un target de vacuna atractivo porque provoca anticuerpos
funcionalmente relevantes de alta calidad, al tiempo que evita el riesgo
potencial de ADE, que generalmente se piensa que está mediado por nAbs
débiles o no nAbs24,30,104.
Por ejemplo, los anticuerpos que se dirigen a un epítopo (S597-603)
cadena abajo del extremo C del SARS-CoV RBD intensificaron notablemente
la infección por SARS-CoV 36,105. El RBD también
contiene epítopos para las respuestas de las células T, como se muestra
en estudios de SARS-CoV, MERS-CoV y SARS-CoV-2 74,106-108. Se
han descrito antígenos basados en RBD en estudios previos para el
desarrollo de vacunas contra el SARS-CoV y MERS-CoV
74,109-111.
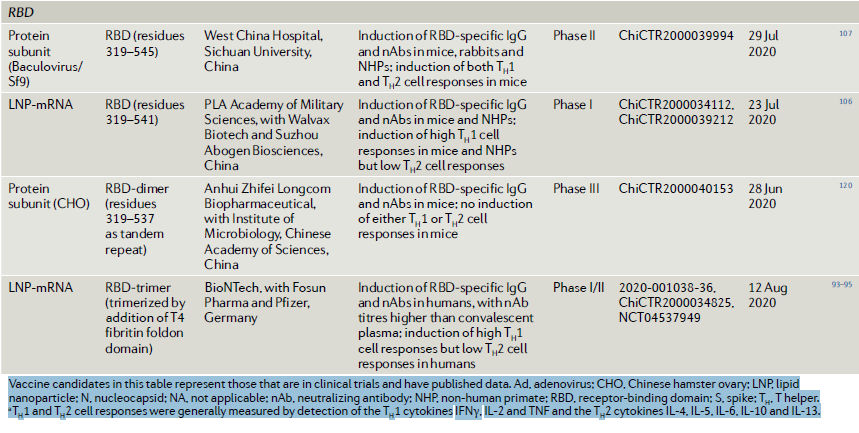
Hasta la fecha, varias vacunas basadas en RBD para COVID-19 han entrado
en ensayos clínicos. Yang y col. reportaron de una vacuna
candidata a COVID-19 basada en RBD generada utilizando una estrategia de
subunidades de proteínas107 (Tabla
1). Esta vacuna indujo nAbs en
ratones, conejos y NHP, y protegió a los NHP contra el desafío con SARS-CoV-2.
Además, demostraron que los sueros inmunes, a diferencia de las células
T esplénicas, desempeñaban un papel protector en los ratones107.
En consonancia con esto, un informe reciente de una vacuna de ADN basada
en RBD también mostró que los nAb, y no las células T inducidas, son los
correlatos inmunitarios de la protección contra COVID-19 en los NHPs
3. En China se está desarrollando una vacuna de ARNm basada
en RBD y actualmente se encuentra en ensayos de fase I.
Esta vacuna candidata, ARCoV, que expresa SARS-CoV RBD administrado por
nanopartículas lipídicas, indujo tanto la producción de nAb como
respuestas sesgadas por células TH1 en modelos de ratón y NHP. La vacuna
protegió a los ratones contra el desafío con una cepa de SARS-CoV-2
adaptada al ratón106.
Sin embargo, el uso de RBD en vacunas se ve comprometido por su
inmunogenicidad limitada debido a su pequeño tamaño molecular y posibles
formas mixtas de múltiples complejos (como monómeros, dímeros o
trímeros). Las estrategias para superar estos inconvenientes incluyen
aumentar el tamaño del antígeno (por ejemplo, fusionando el RBD con un
dominio Fc112-16) o mediante la multimerización del RBD (por
ejemplo, mostrando múltiples copias de RBD en partículas117-119).
Recientemente, para abordar estas limitaciones, nuestro equipo describió
una estrategia generalizable para diseñar una forma dimérica del RBD de
los antígenos beta-CoV adecuada para su uso contra el SARS-CoV-2, MERS-CoV
y SARS-CoV120 (Figura
1)
Los RBD de SARS-CoV, MERS-CoV o SARS-CoV-2 forman espontáneamente dímeros en solución120-124. Los análisis estructurales mostraron, para MERS-CoV118 y SARS-CoV121, que ambos protómeros RBD en un dímero se apilan uno encima del otro a través de los subdominios centrales y exponen el RBM, el sitio principal reconocido por nAbs, lo que indica estructuras de dímero RBD similares para otros CoVs. El diseño guiado por la estructura produjo dímeros RBD homogéneos como una sola cadena repetida en tándem. El antígeno dímero RBD indujo títulos de nAb de 10 a 100 veces más altos que el monómero RBD convencional y fue protector en un modelo de ratón120. Desarrollamos un candidato a vacuna COVID-19, ZF2001, que comprende el dímero RBD como objetivo. Esta vacuna de subunidad proteica se está evaluando actualmente en ensayos clínicos de fase III (Tabla 1).
Además del dímero RBD, se reportó una vacuna de ARNm, BNT162b1 (BioNTech / Pfizer), que expresaba un trímero RBD estabilizado por el dominio de trimerización de pliegue (Figura 1). Los estudios de fase I / II de esta vacuna mostraron de manera alentadora que dos dosis de la vacuna indujeron nAbs a niveles más altos que los de los pacientes convalecientes, además de inducir respuestas sesgadas por las células TH 194,95 (Tabla 1). De hecho, se supone que los antígenos multivalentes permitirían la reticulación de los receptores de las células B para una mejor activación de las células B125. Interesantemente , un estudio reciente describió una candidata a vacuna que comprende múltiples copias del RBD del SARS-CoV-2 que se muestran en matrices en nanopartículas que inducen una relación de unión a nAb marcadamente más baja que una vacuna basada en S-2P119, lo que indica que las respuestas humorales se centran en epítopos reconocidos por nAbs para las vacunas basadas en RBD, que se cree que tienen un potencial de ADE más bajo.
S1- NTD y Subuinidad S2
El S1-NTD también contiene epítopos para nAbs de CoV que se encuentran en pacientes infectados49,63,70,71,126,127 y se ha considerado un objetivo potencial en las vacunas de CoV. Los nAb dirigidos a los NTD generalmente no bloquean directamente la unión al receptor, sino que interfieren con la unión al receptor126,127 o restringen los cambios conformacionales de la proteína S requeridos para la transición de pre-fusión a post-fusión49,127. Es notable que los nAb dirigidos a las NTD de SARS-CoV-2 generalmente exhiben una potencia neutralizante más baja que los nAb específicos de RBD49. Anteriormente informamos sobre una vacuna basada en NTD contra MERS-CoV128. La vacunación con proteína NTD provocó nAbs y respuestas de células T específicas de NTD. Además, redujo las anomalías pulmonares en un modelo de ratón de exposición a MERS-CoV, aunque la inmunogenicidad y la eficacia protectora de la proteína NTD fueron más débiles que la proteína RBD128. La inclusión de NTD en una vacuna COVID-19 ampliaría los epítopos neutralizantes y reduciría el potencial de escape viral de la inmunidad del huésped. Sin embargo, hasta ahora, no se han reportado vacunas contra COVID-19 basadas en NTD
Para la subunidad S2, se han descrito péptidos derivados de HR1 o HR2 de la subunidad S2 de SARS-CoV, MERS-CoV y SARS-CoV-2 que inhiben la fusión viral con células diana y por tanto previenen la infección por virus. Además, se ha informado que los nAbs se dirigen a la subunidad S2 de los CoV, incluido el SARS-CoV-2 49,129-131, lo que sugiere que la subunidad S2 es un target de la vacuna COVID-19. Sin embargo, la subunidad S2 proximal a la membrana contiene un blindaje de N-glucanos más extenso132,133 y es menos accesible para el reconocimiento inmunológico que la subunidad S1 y, por lo tanto, es menos inmunogénica. Los conejos inmunizados con la proteína S2 del SARS-CoV-2 mostraron títulos de nAb mucho más bajos que los inmunizados con la subunidad S1 o las proteínas RBD134. Los anticuerpos dirigidos a la subunidad S2 aislados de pacientes convalecientes mostraron actividades neutralizantes más débiles contra el SARS-CoV-2 que los anticuerpos dirigidos contra el RBD64. Estos estudios sugieren que la subunidad S2 por sí sola puede no ser un objetivo eficaz para las respuestas humorales. Sin embargo, debido a la conservación de la secuencia relativa de la subunidad S2 entre las especies de virus, la subunidad S2 está dirigida por anticuerpos de reacción cruzada y linfocitos T CD4 + que reconocen tanto el SARS-CoV-2 como otros CoV humanos64,131,135,136, lo que sugiere un objetivo potencial en el ámbito universal. Vacunas CoV.
M, E and N proteins.
A diferencia de la proteína S, las proteínas CoV M y E son poco inmunogénicas para las respuestas humorales, presumiblemente debido a sus pequeños ectodominios para el reconocimiento de células inmunitarias y a los pequeños tamaños moleculares1,137 (Figura 1). La transferencia adoptiva de sueros de donantes inmunizados con un vector de virus que expresa proteína M o E no protegió a los ratones contra la infección por SARS-CoV-216. Por lo tanto, las proteínas M y E nunca se han explorado como dianas de vacunas solas contra el SARS-CoV-2 u otros CoV. No obstante, la identidad de secuencia de las proteínas M o E entre el SARS-CoV, MERS-CoV y el SARS-CoV-2 es mucho mayor que para la proteína S y RBD, lo que sugiere el potencial de las proteínas M y E como target para células T con reactividad cruzada. De hecho, se han identificado varios epítopos de células T en las proteínas M y E en estudios previos de inmunidad al SARS-CoV y MERS-CoV74. En este sentido, las proteínas M y E pueden ayudar a ampliar la respuesta de las células T y mejorar la protección cruzada si se incluyen en una vacuna contra el SARS-CoV-2.
La proteína N es la proteína viral más abundante y es altamente inmunogénica durante las infecciones por CoV139. Es un objetivo importante para las respuestas de anticuerpos y también contiene epítopos de células T140. Se informó que los anticuerpos N-específicos protegen a los ratones contra el virus de la hepatitis de ratón, un CoV de ratón, mediante funciones efectoras mediadas por Fc141,142. Sin embargo, los sueros anti-N inmunes no protegieron contra la infección por SARS-CoV-2 en un modelo de ratón16. La inmunización con proteína N también puede provocar respuestas de células T CD4 + y CD8 + en ratones143. Se sabe que los epítopos de células T CD8 + específicos de N protegen a los pollos contra la infección por IBV144. Las partículas del replicón del virus de la encefalitis equina venezolana que expresan un epítopo de células T CD4 + específico de N mostraron una protección completa contra la infección por SARS-CoV138. Estas partículas de replicón de virus también conferían protección cruzada parcial contra MERS-CoV debido a la conservación de la secuencia de proteínas entre virus, lo que resultaba en una carga viral reducida138. Sin embargo, su potencial como objetivo de la vacuna CoV se vio socavado en gran medida por los primeros estudios de SARS-CoV que mostraron que las vacunas que expresan la proteína N no brindan protección y, por el contrario, favorecen la neumonía inducida por infección a través del aumento de la infiltración de eosinófilos pulmonares y la predisposición a respuestas célulares TH2 39,41, causando ERD. Por lo tanto, la inclusión de la proteína N en las vacunas de CoV se complica al equilibrar el aclaramiento viral y la inmunopatogénesis y no se ha informado de ninguna vacuna basada en la proteína N para el COVID-19
Virus total como Target
Las vacunas con virus
inactivados y virus vivos atenuados utilizan el virus completo como
targets de la vacuna. Contienen todas las proteínas estructurales
(proteínas S, N, M y E) y las vacunas de virus vivos atenuados también
pueden generar proteínas no estructurales y accesorias in vivo. Por lo
tanto, estas vacunas candidatas pueden inducir respuestas de anticuerpos
y células T más amplias que las vacunas mencionadas anteriormente, que
se basan en una sola proteína o fragmentos de proteína.
Tres vacunas candidatas (todas desarrolladas en China) basadas en una
estrategia de virus inactivado se encuentran actualmente en ensayos
clínicos de fase III y otra se encuentra en fase I / II (Tabla
1). En un estudio preclínico,
los ratones inmunizados con una vacuna de virus inactivado, PiCoVacc,
obtuvieron anticuerpos específicos de la proteína S (incluidos los
específicos de RBD) y específicos de N5. PiCoVacc y otra
vacuna de virus inactivado, BBIBP-CorV, provocaron una producción
sustancial de nAb en los NHP, pero no indujeron respuestas de células T5,6.
Además, protegieron a los NHP contra el desafío del SARS-CoV-2, lo que
sugiere un papel importante para las respuestas humorales como
correlatos inmunitarios de protección. Los estudios de ensayos clínicos
iniciales de estas dos vacunas candidatas inactivadas mostraron que, al
igual que en los NHP, estas vacunas provocaron sustancial nAbs en
humanos145,146.
Estos candidatos a vacunas inactivadas no indujeron citocinas asociadas a células TH1 ni TH2 en los vacunados. Dado que la captación de componentes de vacunas inactivadas por las células huésped es muy limitada o insignificante, el procesamiento de antígenos para la presentación de péptidos a las células T es ineficaz y puede explicar por qué estas vacunas generalmente no provocan respuestas de las células T.
Además, todas estas vacunas
inactivadas utilizan aluminio como adyuvante, que generalmente es un mal
estimulador de las respuestas de las células T (Tabla 1).
Es destacable que aunque la intensificación de la enfermedad se ha
asociado con vacunas inactivadas contra el SRAS-CoV en modelos animales38,40,
no se ha informado de tal efecto para las vacunas inactivadas para el
SRAS-CoV-2 en estudios preclínicos o clínicos.
Se han autorizado vacunas de virus vivos atenuados para animales contra
IBV, TGEV, BCoV y FIPV140. Sin embargo, se ha observado una
disminución de la inmunidad después de las vacunas contra el IBV y el
BCoV140, lo que genera preocupaciones sobre la duración de
las respuestas inmunitarias provocadas por las vacunas contra el SARS-CoV-2.
De hecho, vale la pena señalar un estudio que muestra que los títulos de nAb en el 93,3% de los pacientes con COVID-19 disminuyeron gradualmente durante el período de estudio de 3 meses147. La relación entre la memoria inmunológica deteriorada y las proteínas CoV en el virus vivo necesita ser explorada más a fondo
El proceso de atenuación del virus para estas vacunas suele llevar mucho tiempo. Hasta el momento, solo tres vacunas vivas atenuadas contra COVID-19 se encuentran en evaluación preclínica, y la desoptimización de codones se utiliza como estrategia de atenuación (OMS)
Observaciones finales
Se esperaría que un target
ideal de la vacuna COVID-19 induzca títulos altos de nAbs, reduzca la
producción de no nAb para minimizar el potencial de ADE, provoque
respuestas robustas con sesgo de células TH1 pero respuestas bajas con
sesgo de células TH2 para reducir el potencial de ERD manteniendo
memoria inmunológica duradera y proporcionando protección cruzada entre
CoV.
Aún no se ha probado ninguna combinación de diferentes targets como vacuna de dianas múltiples, pero podría valer la pena considerar esta posibilidad en el futuro. Hasta la fecha, varias vacunas candidatas han alcanzado las etapas finales de seguridad y eficacia de protección de la vacuna en ensayos clínicos a gran escala, con anuncios muy recientes de BioNTech / Pfizer y Moderna / NIAID que afirman seguridad y niveles muy altos de eficacia de protección para su vacuna candidata de ARNm . Vale la pena señalar que actualmente es difícil comparar las diversas vacunas ya que no existen ensayos estandarizados para la neutralización y estudios de desafío..
Los pros y contras de estos candidatas a vacunas con los respectivos targets objetivos y estrategias deberán analizarse más a fondo para comprender mejor su seguridad, inmunogenicidad y tasa de protección. Además, se necesitarán estudios longitudinales cuidadosos para determinar la duración de cualquier inmunidad protectora y el potencial facilitamiento de la enfermedad para cada vacuna candidata148.
Además, los programas de inmunización requieren una mayor exploración. Por ejemplo, casi todos los candidatos a vacunas en los ensayos clínicos se administran por vía intramuscular o intradérmica. No está claro si los nAbs en el suero llegan al sistema respiratorio, especialmente a los pulmones, para funcionar. Es posible que la vacunación de la mucosa a través del sistema respiratorio sea beneficiosa para inducir respuestas inmunitarias en la mucosa y proteger contra la transmisión del SARS-CoV-2 in situ a través del tracto respiratorio. En consecuencia, un informe reciente describe una vacuna de ChAd intranasal que proporciona principalmente inmunidad esterilizante a la infección por SARS-CoV-2 en un modelo de ratón149. En general, dado que las vacunas COVID-19 serán los primeros esfuerzos de este tipo para una estrategia de control de CoV humano, quedan más desafíos por delante.

Referencias
-
Masters S. P. & Perlman S. in Fields virology 6th edn Ch. 28 (eds Knipe, D. M. & Howley, P. M.). 825–858 (Wolters Kluwer Health/Lippincott Williams & Wilkins, 2013).
-
Tizard, I. R. Vaccination against coronaviruses in domestic animals. Vaccine 38, 5123–5130 (2020).
-
Yu, J. et al. DNA vaccine protection against SARSCoV- 2 in rhesus macaques. Science 369, 806–811 (2020).
-
Mercado, N. B. et al. Single- shot Ad26 vaccine protects against SARS- CoV-2 in rhesus macaques. Nature 586, 583–588 (2020).
-
Gao, Q. et al. Development of an inactivated vaccine candidate for SARS- CoV-2. Science 369, 77–81 (2020).
-
Wang, H. et al. Development of an inactivated vaccine candidate, BBIBP- CorV, with potent protection against SARS- CoV-2. Cell 182, 713–721 (2020).
-
Bloch, E. M. et al. Deployment of convalescent plasma for the prevention and treatment of COVID-19. J. Clin. Invest. 130, 2757–2765 (2020).
-
Shen, C. et al. Treatment of 5 critically ill patients with COVID-19 with convalescent plasma. JAMA 323, 1582–1589 (2020).
-
Fung, M. et al. Treatment of immunocompromised COVID-19 patients with convalescent plasma. Transpl. Infect. Dis. https://doi.org/10.1111/tid.13477 (2020).
-
Hueso, T. et al. Convalescent plasma therapy for B- cell depleted patients with protracted COVID-19 disease. Blood 136, 2290–2295 (2020).
-
Liu, X., Cao, W. & Li, T. High- dose intravenous immunoglobulins in the treatment of severe acute viral pneumonia: the known mechanisms and clinical effects. Front. Immunol. 11, 1660 (2020).
-
Burrage, D. R., Koushesh, S. & Sofat, N. Immunomodulatory drugs in the management of SARS- CoV-2. Front. Immunol. 11, 1844 (2020).
-
Chen, P. et al. SARS- CoV-2 neutralizing antibody LY- CoV555 in outpatients with Covid-19. N. Engl. J. Med. https://doi.org/10.1056/NEJMoa2029849(2020).
-
Mahase, E. Covid-19: FDA authorises neutralising antibody bamlanivimab for non- admitted patients. BMJ 371, m4362 (2020).
-
Addetia, A. et al. Neutralizing antibodies correlate with protection from SARS- CoV-2 in humans during a fishery vessel outbreak with high attack rate. J. Clin. Microbiol. 58, e02107-20 (2020).
-
Sun, J. et al. Generation of a broadly useful model for COVID-19 pathogenesis, vaccination, and treatment. Cell 182, 734–743 (2020).
-
Chen, J. et al. Cellular immune responses to severe acute respiratory syndrome coronavirus (SARS- CoV) infection in senescent BALB/c mice: CD4+ T cells are important in control of SARS- CoV infection. J. Virol. 84, 1289–1301 (2010).
-
Zhao, J., Zhao, J. & Perlman, S. T cell responses are required for protection from clinical disease and for virus clearance in severe acute respiratory syndrome
coronavirus- infected mice. J. Virol. 84, 9318–9325 (2010). -
Zhao, J. et al. Rapid generation of a mouse model for Middle East respiratory syndrome. Proc. Natl Acad. Sci. USA 111, 4970–4975 (2014).
-
Zhu, L. et al. Single- cell sequencing of peripheral mononuclear cells reveals distinct immune response landscapes of COVID-19 and influenza patients. Immunity 53, 685–696 (2020).
-
Mathew, D. et al. Deep immune profiling of COVID-19 patients reveals distinct immunotypes with therapeutic implications. Science 369, eabc851 (2020).
-
Sekine, T. et al. Robust T cell immunity in convalescent individuals with asymptomatic or mild COVID-19. Cell 183, 158–168 (2020).
-
Lee, W. S., Wheatley, A. K., Kent, S. J. & DeKosky, B. J. Antibody- dependent enhancement and SARS- CoV-2 vaccines and therapies. Nat. Microbiol. 5, 1185– 1191 (2020).
-
Rey, F. A., Stiasny, K., Vaney, M. C., Dellarole, M. & Heinz, F. X. The bright and the dark side of human antibody responses to flaviviruses: lessons for vaccine design. EMBO Rep. 19, 206–224 (2018).
-
Halstead, S. B. & O’Rourke, E. J. Dengue viruses and mononuclear phagocytes. I. Infection enhancement by non- neutralizing antibody. J. Exp. Med. 146, 201– 217 (1977).
-
Halstead, S. B., O’Rourke, E. J. & Allison, A. C. Dengue viruses and mononuclear phagocytes. II. Identity of blood and tissue leukocytes supporting in vitro infection. J. Exp. Med. 146, 218–229 (1977).
-
Katzelnick, L. C. et al. Zika virus infection enhances future risk of severe dengue disease. Science 369, 1123–1128 (2020).
-
Takano, T., Kawakami, C., Yamada, S., Satoh, R. & Hohdatsu, T. Antibody- dependent enhancement occurs upon re- infection with the identical serotype virus in feline infectious peritonitis virus infection. J. Vet. Med. Sci. 70, 1315–1321 (2008).
-
Takano, T., Yamada, S., Doki, T. & Hohdatsu, T. Pathogenesis of oral type I feline infectious peritonitis virus (FIPV) infection: antibody- dependent enhancement infection of cats with type I FIPV via the oral route. J. Vet. Med. Sci. 81, 911–915 (2019).
-
Hohdatsu, T., Nakamura, M., Ishizuka, Y., Yamada, H. & Koyama, H. A study on the mechanism of antibodydependent enhancement of feline infectious peritonitis virus infection in feline macrophages by monoclonal antibodies. Arch. Virol. 120, 207–217 (1991).
-
Vennema, H. et al. Early death after feline infectious peritonitis virus challenge due to recombinant vaccinia virus immunization. J. Virol. 64, 1407–1409 (1990).
-
Jaume, M. et al. Anti- severe acute respiratory syndrome coronavirus spike antibodies trigger infection of human immune cells via a pH- and cysteine protease- independent FcγR pathway. J. Virol. 85, 10582–10597 (2011).
-
Kam, Y. W. et al. Antibodies against trimeric S glycoprotein protect hamsters against SARS- CoV challenge despite their capacity to mediate FcγRIIdependent
entry into B cells in vitro. Vaccine 25, 729–740 (2007). -
Wan, Y. et al. Molecular mechanism for antibodydependent enhancement of coronavirus entry. J. Virol. 94, e02015-19 (2020).
-
Wang, S. F. et al. Antibody- dependent SARS coronavirus infection is mediated by antibodies against spike proteins. Biochem. Biophys. Res. Commun. 451, 208–214 (2014).
-
Wang, Q. et al. Immunodominant SARS coronavirus epitopes in humans elicited both enhancing and neutralizing effects on infection in non- human
primates. ACS Infect. Dis. 2, 361–376 (2016). -
Liu, L. et al. Anti- spike IgG causes severe acute lung injury by skewing macrophage responses during acute SARS- CoV infection. JCI Insight 4, e123158 (2019).
-
Tseng, C. T. et al. Immunization with SARS coronavirus vaccines leads to pulmonary immunopathology on challenge with the SARS virus. PLoS ONE 7, e35421 (2012).
-
Deming, D. et al. Vaccine efficacy in senescent mice challenged with recombinant SARS- CoV bearing epidemic and zoonotic spike variants. PLoS Med. 3,
e525 (2006). -
Bolles, M. et al. A double- inactivated severe acute respiratory syndrome coronavirus vaccine provides incomplete protection in mice and induces increased
eosinophilic proinflammatory pulmonary response upon challenge. J. Virol. 85, 12201–12215 (2011). -
Yasui, F. et al. Prior immunization with severe acute respiratory syndrome (SARS)-associated coronavirus (SARS- CoV) nucleocapsid protein causes severe
pneumonia in mice infected with SARS- CoV. J. Immunol. 181, 6337–6348 (2008). -
Luo, F. et al. Evaluation of antibody- dependent enhancement of SARS- CoV infection in rhesus macaques immunized with an inactivated SARS- CoV
vaccine. Virol. Sin. 33, 201–204 (2018). -
Qin, E. et al. Immunogenicity and protective efficacy in monkeys of purified inactivated Vero- cell SARS vaccine. Vaccine 24, 1028–1034 (2006).
-
Graham, B. S. Rapid COVID-19 vaccine development. Science 368, 945–946 (2020).
-
Srinivasan, S. et al. Structural genomics of SARSCoV- 2 indicates evolutionary conserved functional regions of viral proteins. Viruses 12, 360 (2020).
-
Yuan, Y. et al. Cryo- EM structures of MERS- CoV and SARS- CoV spike glycoproteins reveal the dynamic receptor binding domains. Nat. Commun. 8, 15092 (2017).
-
Chen, Q. et al. A report of clinical diagnosis and treatment of nine cases of coronavirus disease 2019. J. Med. Virol. 92, 683–687 (2020).
-
Wrapp, D. et al. Cryo- EM structure of the 2019-nCoV spike in the prefusion conformation. Science 367, 1260–1263 (2020).
-
Chi, X. et al. A neutralizing human antibody binds to the N- terminal domain of the Spike protein of SARS- CoV-2. Science 369, 650–655 (2020).
-
Lu, G., Wang, Q. & Gao, G. F. Bat- to-human: spike features determining ‘host jump’ of coronaviruses SARS- CoV, MERS- CoV, and beyond. Trends Microbiol.
23, 468–478 (2015). -
Li, F. Receptor recognition mechanisms of coronaviruses: a decade of structural studies. J. Virol. 89, 1954–1964 (2015).
-
Cai, Y. et al. Distinct conformational states of SARSCoV- 2 spike protein. Science 369, 1586–1592 (2020).
-
Gao, G. F. in Combating the Threat of Pandemic Influenza: Drug Discovery Approaches (eds Torrence, P. F.) 226–246 (John Wiley & Sons, 2007).
-
Premkumar, L. et al. The receptor binding domain of the viral spike protein is an immunodominant and highly specific target of antibodies in SARS- CoV-2
patients. Sci. Immunol. 5, eabc8413 (2020). -
Wan, J. et al. Human- IgG-neutralizing monoclonal antibodies block the SARS- CoV-2 infection. Cell Rep. 32, 107918 (2020).
-
Yuan, M., Liu, H., Wu, N. C. & Wilson, I. A. Recognition of the SARS- CoV-2 receptor binding domain by neutralizing antibodies. Biochem. Biophys. Res. Commun. https://doi.org/10.1016/j.bbrc.2020.10.012 (2020).
-
Robbiani, D. F. et al. Convergent antibody responses to SARS- CoV-2 in convalescent individuals. Nature 584, 437–442 (2020).
-
Ju, B. et al. Human neutralizing antibodies elicited by SARS- CoV-2 infection. Nature 584, 115–119 (2020).
-
Shi, R. et al. A human neutralizing antibody targets the receptor- binding site of SARS- CoV-2. Nature 584, 120–124 (2020).
-
Du, S. et al. Structurally resolved SARS- CoV-2 antibody shows high efficacy in severely infected hamsters and provides a potent cocktail pairing
strategy. Cell 183, 1013–1023 (2020). -
Zost, S. J. et al. Potently neutralizing and protective human antibodies against SARS- CoV-2. Nature 584, 443–449 (2020).
-
Wu, Y. et al. A noncompeting pair of human neutralizing antibodies block COVID-19 virus binding to its receptor ACE2. Science 368, 1274–1278 (2020).
-
Brouwer, P. J. M. et al. Potent neutralizing antibodies from COVID-19 patients define multiple targets of vulnerability. Science 369, 643–650 (2020).
-
Wec, A. Z. et al. Broad neutralization of SARS- related viruses by human monoclonal antibodies. Science 369, 731–736 (2020).
-
Rogers, T. F. et al. Isolation of potent SARS- CoV-2 neutralizing antibodies and protection from disease in a small animal model. Science 369, 956–963 (2020).
-
Hansen, J. et al. Studies in humanized mice and convalescent humans yield a SARS- CoV-2 antibody cocktail. Science 369, 1010–1014 (2020).
-
Guo, Z. D. et al. Aerosol and surface distribution of severe acute respiratory syndrome coronavirus 2 in hospital wards, Wuhan, China, 2020. Emerg. Infect.
Dis. 26, 1583–1591 (2020). -
Barnes, C. O. et al. Structures of human antibodies bound to SARS- CoV-2 spike reveal common epitopes and recurrent features of antibodies. Cell 182, 828–842 (2020).
-
Cao, Y. et al. Potent neutralizing antibodies against SARS- CoV-2 identified by high- throughput single- cell sequencing of convalescent patients’ B cells. Cell 182, 73–84 (2020).
-
Liu, L. et al. Potent neutralizing antibodies against multiple epitopes on SARS- CoV-2 spike. Nature 584, 450–456 (2020).
-
Jiang, S., Zhang, X. & Du, L. Therapeutic antibodies and fusion inhibitors targeting the spike protein of SARS- CoV-2. Expert Opin. Ther. Targets https://doi.org/ 10.1080/14728222.2020.1820482 (2020).
-
Zhu, J. et al. Following the rule: formation of the 6-helix bundle of the fusion core from severe acute respiratory syndrome coronavirus spike protein and identification of potent peptide inhibitors. Biochem. Biophys. Res. Commun. 319, 283–288 (2004).
-
Sun, H. et al. Structural basis of HCoV-19 fusion core and an effective inhibition peptide against virus entry. Emerg. Microbes Infect. 9, 1238–1241 (2020).
-
Liu, W. J. et al. T- cell immunity of SARS- CoV: Implications for vaccine development against MERS- CoV. Antivir. Res. 137, 82–92 (2017).
-
Grifoni, A. et al. Targets of T cell responses to SARSCoV- 2 coronavirus in humans with COVID-19 disease and unexposed individuals. Cell 181, 1489–1501
(2020). -
Zhu, F. C. et al. Immunogenicity and safety of a recombinant adenovirus type-5-vectored COVID-19 vaccine in healthy adults aged 18 years or older: a randomised, double- blind, placebo- controlled, phase 2 trial. Lancet 396, 479–488 (2020).
-
Zhu, F. C. et al. Safety, tolerability, and immunogenicity of a recombinant adenovirus type-5 vectored COVID-19 vaccine: a dose- escalation, open- label, non- randomised, first- in-human trial. Lancet 395, 1845–1854 (2020).
-
van Doremalen, N. et al. ChAdOx1 nCoV-19 vaccine prevents SARS- CoV-2 pneumonia in rhesus macaques. Nature 586, 578–582 (2020).
-
79. Folegatti, P. M. et al. Safety and immunogenicity of the ChAdOx1 nCoV-19 vaccine against SARS- CoV-2: a preliminary report of a phase 1/2, single- blind, randomised controlled trial. Lancet 396, 467–478 (2020).
-
Logunov, D. Y. et al. Safety and immunogenicity of an rAd26 and rAd5 vector- based heterologous primeboost COVID-19 vaccine in two formulations: two open, non- randomised phase 1/2 studies from Russia. Lancet 396, 887–897 (2020).
-
Smith, T. R. F. et al. Immunogenicity of a DNA vaccine candidate for COVID-19. Nat. Commun. 11, 2601 (2020).
-
Ma, J. et al. Cryo- EM structure of S- Trimer, a subunit vaccine candidate for COVID-19. Preprint at bioRxiv https://doi.org/10.1101/2020.09.21.306357 (2020).
-
Sanders, R. W. & Moore, J. P. Native- like Env trimers as a platform for HIV-1 vaccine design. Immunol. Rev. 275, 161–182 (2017).
-
Liang, J. G. et al. S- Trimer, a COVID-19 subunit vaccine candidate, induces protective immunity in nonhuman primates. Preprint at bioRxiv https://doi.org/10.1101/ 2020.09.24.311027 (2020).
-
Pallesen, J. et al. Immunogenicity and structures of a rationally designed prefusion MERS- CoV spike antigen. Proc. Natl Acad. Sci. USA 114, E7348–E7357 (2017).
-
Corbett, K. S. et al. Evaluation of the mRNA-1273 vaccine against SARS- CoV-2 in nonhuman primates. N. Engl. J. Med. 383, 1544–1555 (2020).
-
Corbett, K. S. et al. SARS- CoV-2 mRNA vaccine design enabled by prototype pathogen preparedness. Nature 586, 567–571 (2020).
-
Jackson, L. A. et al. An mRNA vaccine against SARSCoV- 2 — preliminary report. N. Engl. J. Med. 383, 1920–1931 (2020).
-
Walsh, E. E. et al. RNA- based COVID-19 vaccine BNT162b2 selected for a pivotal efficacy study. Preprint at medRxiv https://doi.org/10.1101/ 2020.08.17.20176651 (2020).
-
Tian, J.-H. et al. SARS- CoV-2 spike glycoprotein vaccine candidate NVX- CoV2373 elicits immunogenicity in baboons and protection in mice. Preprint at bioRxiv https://doi.org/10.1101/ 2020.06.29.178509 (2020).
-
91. Keech, C. et al. Phase 1-2 trial of a SARS- CoV-2 recombinant spike protein nanoparticle vaccine. N. Engl. J. Med. https://doi.org/10.1056/NEJMoa 2026920 (2020).
-
Tostanoski, L. H. et al. Ad26 vaccine protects against SARS- CoV-2 severe clinical disease in hamsters. Nat. Med. 26, 1694–1700 (2020).
-
Walsh, E. E. et al. Safety and immunogenicity of two RNA- based Covid-19 vaccine candidates. N. Engl. J. Med. https://doi.org/10.1056/NEJMoa2027906 (2020).
-
Mulligan, M. J. et al. Phase 1/2 study of COVID-19 RNA vaccine BNT162b1 in adults. Nature 586, 589–593 (2020).
-
Sahin, U. et al. COVID-19 vaccine BNT162b1 elicits human antibody and TH1 T- cell responses. Nature 586, 594–599 (2020).
-
Bangaru, S. et al. Structural analysis of full- length SARS- CoV-2 spike protein from an advanced vaccine candidate. Science 370, 1089–1094 (2020).
-
Bos, R. et al. Ad26 vector- based COVID-19 vaccine encoding a prefusion- stabilized SARS- CoV-2 Spike immunogen induces potent humoral and cellular
immune responses. NPJ Vaccines 5, 91 (2020). -
Pfizer. Pfizer and BioNTech announce vaccine candidate against COVID-19 achieved success in first interim analysis from phase 3 study. Pfizer https:// www.pfizer.com/news/press- release/press- releasedetail/pfizer- and-biontech- announce-vaccinecandidate-against (2020).
-
Moderna. Moderna’s COVID-19 vaccine candidate meets its primary efficacy endpoint in the first interim analysis of the phase 3 COVE study. Moderna https://investors.modernatx.com/node/10316/pdf (2020).
-
Mahase, E. Covid-19: UK approves Pfizer and BioNTech vaccine with rollout due to start next week. BMJ 371, m4714 (2020).
-
Hsieh, C. L. et al. Structure- based design of prefusion- stabilized SARS- CoV-2 spikes. Science 369, 1501–1505 (2020).
-
Lu, G. et al. Molecular basis of binding between novel human coronavirus MERS- CoV and its receptor CD26. Nature 500, 227–231 (2013).
-
Grant, O. C., Montgomery, D., Ito, K. & Woods, R. J. Analysis of the SARS- CoV-2 spike protein glycan shield reveals implications for immune recognition. Sci. Rep. 10, 14991 (2020).
-
Weiss, R. C. & Scott, F. W. Antibody- mediated enhancement of disease in feline infectious peritonitis: comparisons with dengue hemorrhagic fever. Comp. Immunol. Microbiol. Infect. Dis. 4, 175–189 (1981).
-
105. Su, S., Du, L. & Jiang, S. Learning from the past: development of safe and effective COVID-19 vaccines. Nat. Rev. Microbiol. https://doi.org/10.1038/s41579- 020-00462-y (2020).
-
Zhang, N. N. et al. A Thermostable mRNA vaccine against COVID-19. Cell 182, 1271–1283 (2020).
-
Yang, J. et al. A vaccine targeting the RBD of the S protein of SARS- CoV-2 induces protective immunity. Nature 586, 572–577 (2020).
-
Zhou, M. et al. Screening and identification of severe acute respiratory syndrome- associated coronavirusspecific CTL epitopes. J. Immunol. 177, 2138–2145
(2006). -
Jiang, S. et al. Roadmap to developing a recombinant coronavirus S protein receptor- binding domain vaccine for severe acute respiratory syndrome. Expert Rev. Vaccines 11, 1405–1413 (2012).
-
Wang, C. B. Analysis of low positive rate of nucleic acid detection method used for diagnosis of novel coronavirus pneumonia [In Chinese]. Zhonghua Yi Xue
Za Zhi 100, E010 (2020). -
Zhou, Y., Yang, Y., Huang, J., Jiang, S. & Du, L. Advances in MERS- CoV vaccines and therapeutics based on the receptor- binding domain. Viruses 11, 60 (2019).
-
Gu, H. et al. Adaptation of SARS- CoV-2 in BALB/c mice for testing vaccine efficacy. Science 369, 1603–1607 (2020).
-
Du, L. et al. Receptor- binding domain of SARS- CoV spike protein induces long- term protective immunity in an animal model. Vaccine 25, 2832–2838 (2007).
-
Du, L. et al. A truncated receptor- binding domain of MERS- CoV spike protein potently inhibits MERS- CoV infection and induces strong neutralizing antibody responses: implication for developing therapeutics and vaccines. PLoS ONE 8, e81587 (2013).
-
Ma, C. et al. Searching for an ideal vaccine candidate among different MERS coronavirus receptor- binding fragments–the importance of immunofocusing in subunit vaccine design. Vaccine 32, 6170–6176 (2014).
-
He, Y. et al. Receptor- binding domain of SARS- CoV spike protein induces highly potent neutralizing antibodies: implication for developing subunit vaccine. Biochem. Biophys. Res. Commun. 324, 773–781 (2004).
-
Wang, C. et al. Novel chimeric virus- like particles vaccine displaying MERS- CoV receptor- binding domain induce specific humoral and cellular immune response in mice. Antivir. Res. 140, 55–61 (2017).
-
Kim, Y. S. et al. Chaperna- mediated assembly of ferritin- based middle east respiratory syndromecoronavirus nanoparticles. Front. Immunol. 9, 1093 (2018).
-
Walls, A. C. et al. Elicitation of potent neutralizing antibody responses by designed protein nanoparticle vaccines for SARS- CoV-2. Cell 183, 1367–1382 (2020).
-
Dai, L. et al. A universal design of betacoronavirus vaccines against COVID-19, MERS, and SARS. Cell 182, 722–733 (2020).
-
Hwang, W. C. et al. Structural basis of neutralization by a human anti- severe acute respiratory syndrome spike protein antibody, 80R. J. Biol. Chem. 281, 34610–34616 (2006).
-
Lan, J. et al. Structure of the SARS- CoV-2 spike receptor- binding domain bound to the ACE2 receptor. Nature 581, 215–220 (2020).
-
Xiao, X., Feng, Y., Chakraborti, S. & Dimitrov, D. S. Oligomerization of the SARS- CoV S glycoprotein: dimerization of the N- terminus and trimerization of the ectodomain. Biochem. Biophys. Res. Commun. 322, 93–99 (2004).
-
Zhang, S. et al. Structural definition of a unique neutralization epitope on the receptor- binding domain of MERS- CoV spike glycoprotein. Cell Rep. 24, 441–452 (2018).
-
Bachmann, M. F. & Zinkernagel, R. M. Neutralizing antiviral B cell responses. Annu. Rev. Immunol. 15, 235–270 (1997).
-
Wang, N. et al. Structural definition of a neutralizationsensitive epitope on the MERS- CoV S1-NTD. Cell Rep. 28, 3395–3405 (2019).
-
Zhou, H. et al. Structural definition of a neutralization epitope on the N- terminal domain of MERS- CoV spike glycoprotein. Nat. Commun. 10, 3068 (2019).
-
Jiaming, L. et al. The recombinant N- terminal domain of spike proteins is a potential vaccine against Middle East respiratory syndrome coronavirus (MERS- CoV) infection. Vaccine 35, 10–18 (2017).
-
Huang, Y., Yang, C., Xu, X. F., Xu, W. & Liu, S. W. Structural and functional properties of SARS- CoV-2 spike protein: potential antivirus drug development for COVID-19. Acta Pharmacol. Sin. 41, 1141–1149 (2020).
-
Song, G. et al. Cross- reactive serum and memory B cell responses to spike protein in SARS- CoV-2 and endemic coronavirus infection. Preprint at bioRxiv
https://doi.org/10.1101/2020.09.22.308965(2020). -
Ng, K. W. et al. Preexisting and de novo humoral immunity to SARS- CoV-2 in humans. Science https://doi.org/10.1126/science.abe1107 (2020).
-
Watanabe, Y., Allen, J. D., Wrapp, D., McLellan, J. S. & Crispin, M. Site- specific glycan analysis of the SARS- CoV-2 spike. Science 369, 330–333 (2020).
-
Walls, A. C. et al. Structure, function, and antigenicity of the SARS- CoV-2 spike glycoprotein. Cell 181, 281–292 (2020).
-
Ravichandran, S. et al. Antibody signature induced by SARS- CoV-2 spike protein immunogens in rabbits. Sci. Transl. Med. 12, eabc3539 (2020).
-
Jeyanathan, M. et al. Immunological considerations for COVID-19 vaccine strategies. Nat. Rev. Immunol. 20, 615–632 (2020).
-
Braun, J. et al. SARS- CoV-2-reactive T cells in healthy donors and patients with COVID-19. Nature 587, 270–274 (2020).
-
Du, L., He, Y., Jiang, S. & Zheng, B. J. Development of subunit vaccines against severe acute respiratory syndrome. Drugs Today 44, 63–73 (2008).
-
Zhao, J. et al. Airway memory CD4+ T cells mediate protective immunity against emerging respiratory coronaviruses. Immunity 44, 1379–1391 (2016).
-
Long, Q. X. et al. Antibody responses to SARS- CoV-2 in patients with COVID-19. Nat. Med. 26, 845–848 (2020).
-
Sariol, A. & Perlman, S. Lessons for COVID-19 immunity from other coronavirus infections. Immunity 53, 248–263 (2020).
-
Nakanaga, K., Yamanouchi, K. & Fujiwara, K. Protective effect of monoclonal antibodies on lethal mouse hepatitis virus infection in mice. J. Virol. 59, 168–171 (1986).
-
Lecomte, J. et al. Protection from mouse hepatitis virus type 3-induced acute disease by an antinucleoprotein monoclonal antibody. Brief report.Arch. Virol. 97, 123–130 (1987).
-
Liu, S. J. et al. Immunological characterizations of the nucleocapsid protein based SARS vaccine candidates. Vaccine 24, 3100–3108 (2006).
-
Collisson, E. W., Pei, J., Dzielawa, J. & Seo, S. H. Cytotoxic T lymphocytes are critical in the control of infectious bronchitis virus in poultry. Dev. Comp. Immunol. 24, 187–200 (2000).
-
Zhang, Y. et al. Safety, tolerability, and immunogenicity of an inactivated SARS- CoV-2 vaccine in healthy adults aged 18-59 years: a randomised, double- blind, placebo- controlled, phase 1/2 clinical trial. Lancet Infect. Dis. https://doi.org/10.1016/S1473-3099(20)30843-4 (2020)
-
Xia, S. et al. Effect of an inactivated vaccine against SARS- CoV-2 on safety and immunogenicity outcomes: interim analysis of 2 randomized clinical trials. JAMA 324, 951–960 (2020).
-
Wang, K. et al. Longitudinal dynamics of the neutralizing antibody response to SARS- CoV-2 infection. Clin. Infect. Dis. https://doi.org/10.1093/cid/ciaa1143 (2020).
-
Moore, J. P. & Klasse, P. J. COVID-19 Vaccines: “Warp Speed” needs mind melds, not warped minds. J. Virol. 94, e01083-20 (2020).
-
Hassan, A. O. et al. A single- dose intranasal ChAd vaccine protects upper and lower respiratory tracts against SARS- CoV-2. Cell 183, 169–184 (2020).
-
Xia, S. et al. Safety and immunogenicity of an inactivated SARS- CoV-2 vaccine, BBIBP- CorV: a randomised, double- blind, placebo- controlled, phase 1/2 trial. Lancet Infect. Dis. https://doi.org/ 10.1016/S1473-3099(20)30831-8 (2020).
-
Che, Y. et al. Randomized, double- blinded and placebo- controlled phase II trial of an inactivated SARS- CoV-2 vaccine in healthy adults. Clin. Infect. Dis. https://doi.org/10.1093/cid/ciaa1703 (2020).
-
Ramasamy, M. N. et al. Safety and immunogenicity of ChAdOx1 nCoV-19 vaccine administered in a primeboost regimen in young and old adults (COV002): a single- blind, randomised, controlled, phase 2/3 trial. Lancet https://doi.org/10.1016/S0140-6736(20) 32466-1 (2020).
-
Anderson, E. J. et al. Safety and immunogenicity of SARS-CoV-2 mRNA-1273 vaccine in older adults. N. Engl. J. Med. https://doi.org/10.1056/NEJMoa 2028436 (2020).
-
Guebre- Xabier, M. et al. NVX- CoV2373 vaccine protects cynomolgus macaque upper and lower airways against SARS- CoV-2 challenge. Vaccine 38, 7892–7896 (2020).